Produkte
Lifecosm SARS-Cov-2-RT-PCR-Nachweiskit für 2019-nCoV
Erwartete Nutzung
Dieses Kit dient zum qualitativen Nachweis des neuen Coronavirus (2019-nCoV) mithilfe von Rachenabstrichen, Nasenrachenabstrichen, bronchoalveolärer Lavageflüssigkeit und Sputum. Das Nachweisergebnis dieses Produkts dient nur als klinische Referenz und sollte nicht als einziger Beweis für die klinische Diagnose und Behandlung verwendet werden. Eine umfassende Analyse des Zustands wird in Kombination mit den klinischen Manifestationen des Patienten und anderen Labortests empfohlen.
Prüfprinzip
Das Kit basiert auf der einstufigen RT-PCR-Technologie. Die ORF1ab- und N-Gene des neuen Coronavirus 2019 (2019-nCoV) wurden als Zielregionen für die Amplifikation ausgewählt. Spezifische Primer und Fluoreszenzsonden (N-Gensonden sind mit FAM und ORF1ab-Sonden mit HEX markiert) dienen dem Nachweis der RNA des neuen Coronavirus 2019 in Proben. Das Kit enthält außerdem ein endogenes internes Kontrollsystem (interne Kontrollgensonde mit CY5-Markierung), um den Prozess der Probenentnahme sowie der RNA- und PCR-Amplifikation zu überwachen und so falsch-negative Ergebnisse zu reduzieren.
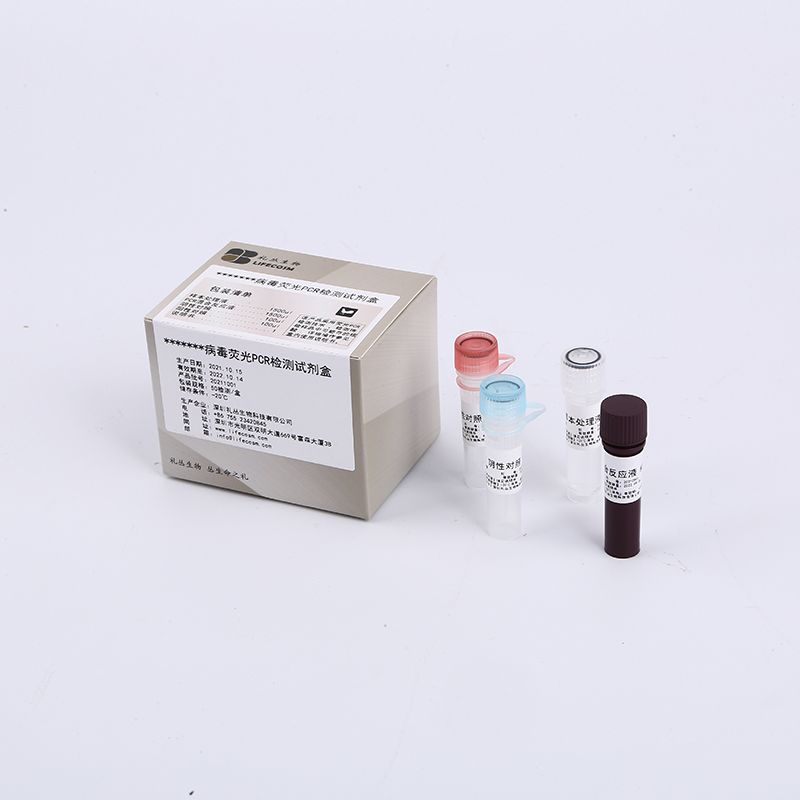
Hauptkomponenten
| Komponenten | Volumen(48T/Kit) |
| RT-PCR-Reaktionslösung | 96 µl |
| nCOV-Primer-TaqMan-Sondenmischung (ORF1ab, N-Gen, RnaseP-Gen) | 864 µl |
| Negativkontrolle | 1500 µl |
| nCOV-Positivkontrolle (ORF1ab-N-Gen) | 1500 µl |
Eigene Reagenzien: RNA-Extraktions- oder Reinigungsreagenzien. Negative/positive Kontrolle: Die positive Kontrolle ist RNA, die das Zielfragment enthält, während die negative Kontrolle nukleinsäurefreies Wasser ist. Bei der Verwendung sollten sie an der Extraktion teilnehmen und als infektiös gelten. Sie sollten gemäß den geltenden Vorschriften gehandhabt und entsorgt werden.
Das interne Referenzgen ist das menschliche RnaseP-Gen.
Lagerbedingungen und Verfallsdatum
-20 ± 5 °C, mehr als 5-maliges Einfrieren und Auftauen vermeiden, 6 Monate gültig.
Anwendbares Instrument
Mit FAM/HEX/CY5 und anderen Mehrkanal-Fluoreszenz-PCR-Instrumenten.
Probenanforderungen
1. Anwendbare Probentypen: Rachenabstriche, Nasopharyngealabstriche, bronchoalveoläre Lavageflüssigkeit, Sputum.
2.Probenentnahme (aseptische Technik)
Rachenabstrich: Mit zwei Tupfern gleichzeitig die Mandeln und die hintere Rachenwand abwischen, anschließend den Tupferkopf in ein Reagenzglas mit der Probenlösung tauchen
Auswurf: Nach einem starken Husten des Patienten wird der ausgehustete Auswurf in einem Reagenzglas mit Schraubverschluss gesammelt, das die Probenlösung enthält; bronchoalveoläre Lavageflüssigkeit: Die Probenentnahme erfolgt durch medizinisches Fachpersonal. 3. Lagerung und Transport der Proben
Proben für die Virusisolierung und RNA-Tests sollten so schnell wie möglich getestet werden. Proben, die innerhalb von 24 Stunden nachweisbar sind, können bei 4 °C gelagert werden; Proben, die innerhalb von 24 Stunden nicht nachweisbar sind,
Stunden sollten bei -70 °C oder darunter gelagert werden (wenn keine Lagerbedingung von -70 °C vorliegt, sollten sie
Zwischenlagerung im Kühlschrank bei -20 °C). Proben sollten während des Transports nicht wiederholt eingefroren und aufgetaut werden. Proben sollten nach der Entnahme so schnell wie möglich an das Labor geschickt werden. Bei längeren Transporten empfiehlt sich die Lagerung auf Trockeneis.
Testmethoden
1 Probenverarbeitung und RNA-Extraktion (Bereich Probenverarbeitung)
Es wird empfohlen, 200 μl flüssige Probe für die RNA-Extraktion zu entnehmen. Die entsprechenden Extraktionsschritte finden Sie in den Anweisungen der kommerziellen RNA-Extraktionskits. Sowohl die negative als auch die negative
Kontrollen in diesem Kit waren an der Extraktion beteiligt.
2 PCR-Reagenzienvorbereitung (Bereich zur Reagenzienvorbereitung)
2.1 Alle Komponenten aus dem Kit entnehmen, bei Raumtemperatur auftauen und mischen. Vor Gebrauch einige Sekunden bei 8.000 U/min zentrifugieren; die benötigte Reagenzienmenge berechnen und das Reaktionssystem wie in der folgenden Tabelle dargestellt vorbereiten:
| Komponenten | N Portion (25µl System) |
| nCOV-Primer-TaqMan-Sondenmischung | 18 µl × N |
| RT-PCR-Reaktionslösung | 2 µl × N |
| *N = Anzahl der getesteten Proben + 1 (Negativkontrolle) + 1 (nCOVpositive Kontrolle) | |
2.2 Nach gründlichem Mischen der Komponenten zentrifugieren Sie kurz, damit die gesamte Flüssigkeit an der Röhrchenwand auf den Boden des Röhrchens sinkt, und geben Sie dann das 20-µl-Amplifikationssystem in das PCR-Röhrchen.
3 Probenahme (Bereich Probenvorbereitung)
Nach der Extraktion werden jeweils 5 μl der Negativ- und Positivkontrolle zugegeben. Die RNA der zu testenden Probe wird in das PCR-Reaktionsgefäß gegeben.
Verschließen Sie das Röhrchen fest und zentrifugieren Sie es einige Sekunden lang bei 8.000 U/min, bevor Sie es in den Amplifikationsdetektionsbereich überführen.
4 PCR-Amplifikation (verstärkter Nachweisbereich)
4.1 Platzieren Sie das Reaktionsgefäß in der Probenzelle des Geräts und stellen Sie die Parameter wie folgt ein:
| Bühne | Zyklus Nummer | Temperatur(°C) | Zeit | SammlungWebsite |
| UmkehrenTranskription | 1 | 42 | 10 Minuten | - |
| Vordenaturierungn | 1 | 95 | 1 Minute | - |
| Zyklus | 45 | 95 | 15 Sekunden | - |
| 60 | 30er Jahre | Datenerfassung |
Auswahl des Geräteerkennungskanals: Wählen Sie den Kanal FAM, HEX oder CY5 für das Fluoreszenzsignal. Für die Referenzfluoreszenz NONE wählen Sie bitte nicht ROX.
5 Ergebnisanalyse (Informationen zur Einstellung finden Sie in der Versuchsanleitung des jeweiligen Instruments.)
Speichern Sie die Ergebnisse nach der Reaktion. Passen Sie nach der Analyse den Startwert, den Endwert und den Schwellenwert der Basislinie entsprechend dem Bild an (der Benutzer kann die Anpassung entsprechend der tatsächlichen Situation vornehmen, der Startwert kann auf 3 bis 15 eingestellt werden, der Endwert kann auf 5 bis 20 eingestellt werden, Anpassung) im logarithmischen Diagramm. Am Schwellenwert des Fensters befindet sich die Schwellenwertlinie in der logarithmischen Phase und die Amplifikationskurve der Negativkontrolle ist eine gerade Linie oder liegt unterhalb der Schwellenwertlinie.
6 Qualitätskontrolle (Eine Verfahrenskontrolle ist im Test enthalten) Negativkontrolle: Keine offensichtliche Amplifikationskurve für FAM-, HEX- und CY5-Erkennungskanäle
COV-Positivkontrolle: deutliche Amplifikationskurve der FAM- und HEX-Erkennungskanäle, Ct-Wert ≤ 32, aber keine Amplifikationskurve des CY5-Kanals;
Die oben genannten Anforderungen müssen gleichzeitig im selben Experiment erfüllt sein, andernfalls ist das Experiment ungültig und muss wiederholt werden.
7 Ermittlung der Ergebnisse.
7.1 Wenn in den FAM- und HEX-Kanälen der Testprobe keine Amplifikationskurve oder kein Ct-Wert > 40 vorliegt und im CY5-Kanal eine Amplifikationskurve vorhanden ist, kann davon ausgegangen werden, dass die Probe keine RNA des neuen Coronavirus 2019 (2019-nCoV) enthält.
.2 Wenn die Testprobe offensichtliche Amplifikationskurven in den FAM- und HEX-Kanälen aufweist und der Ct-Wert ≤40 beträgt, kann davon ausgegangen werden, dass die Probe positiv auf das neue Coronavirus 2019 (2019-nCoV) ist.
7.3 Wenn die Testprobe nur in einem Kanal von FAM oder HEX eine klare Amplifikationskurve aufweist und der Ct-Wert ≤40 ist, während im anderen Kanal keine Amplifikationskurve vorliegt, müssen die Ergebnisse erneut getestet werden. Wenn die Ergebnisse des erneuten Tests konsistent sind, kann die Probe als positiv für die neue
Coronavirus 2019 (2019-nCoV). Wenn das Ergebnis des erneuten Tests negativ ist, kann davon ausgegangen werden, dass die Probe negativ auf das neue Coronavirus 2019 (2019-nCoV) getestet wurde.
Positiver Urteilswert
Mit der ROC-Kurvenmethode wird der CT-Referenzwert des Kits ermittelt, und der interne Kontrollreferenzwert beträgt 40.
Interpretation der Testergebnisse
1.Jedes Experiment sollte auf negative und positive Kontrollen getestet werden. Testergebnisse können nur ermittelt werden, wenn die Kontrollen den Anforderungen der Qualitätskontrolle entsprechen
2.Wenn die FAM- und HEX-Erkennungskanäle positiv sind, kann das Ergebnis des CY5-Kanals (interner Kontrollkanal) aufgrund von Systemkonkurrenz negativ sein.
3. Wenn das interne Kontrollergebnis negativ ist und die FAM- und HEX-Detektionskanäle des Reagenzglases ebenfalls negativ sind, bedeutet dies, dass das System deaktiviert ist oder die Bedienung fehlerhaft ist. Der Test ist ungültig. Daher müssen die Proben erneut getestet werden.